軟件準備
# hisat2安裝
conda install hisat2 -y
hisat
# perl環境報錯
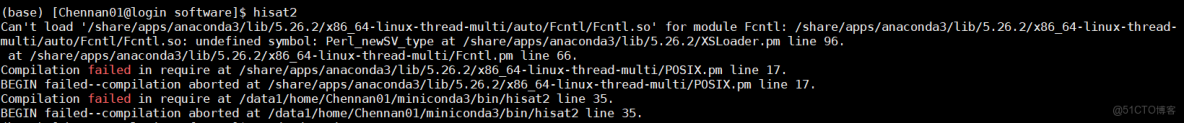
conda install -c conda-forge perl=5.26.2 -y問題:Perl模塊Fcntl的共享庫Fcntl.so無法加載,原因是未定義符號Perl_newSV_type。通常是因為Perl版本不匹配或模塊編譯時使用的Perl與當前運行的Perl不一致。
# 執行hisat2腳本使用的perl版本:【指定miniconda3的perl】
[Chennan01@login software]$ head -1 /data1/home/Chennan01/miniconda3/bin/hisat2
#!/data1/home/Chennan01/miniconda3/bin/perl //明確使用miniconda的Perl
# 執行到特定模塊時,模塊搜索的perl版本:【指定共享目錄的perl】
[Chennan01@login software]$ echo $PERL5LIB
/share/apps/anaconda3/lib/5.26.2: //環境變量PERL5LIB被設置為強制搜索系統路徑
# 查看Perl模塊搜索路徑:當Perl加載Fcntl模塊時,它會按順序搜索這些路徑。【先用共享目錄的perl】
[Chennan01@login software]$ perl -e 'print join("\n", @INC)'
//-e表示"execute"(執行)。允許在命令行中直接嵌入Perl代碼,而無需創建腳本文件。單引號內的內容是Perl代碼
//@INC變量:這是Perl的內置特殊數組,包含Perl搜索模塊(.pm文件)的所有目錄路徑。類似於Python的sys.path或shell的$PATH
//join()是Perl函數,將數組元素連接成字符串。"\n"是換行符,將@INC數組中的每個路徑用換行符連接
//結果:每個路徑佔一行
/share/apps/anaconda3/lib/5.26.2/x86_64-linux-thread-multi
/share/apps/anaconda3/lib/5.26.2
/data1/home/Chennan01/miniconda3/lib/site_perl/5.26.2/x86_64-linux-thread-multi
/data1/home/Chennan01/miniconda3/lib/site_perl/5.26.2
/data1/home/Chennan01/miniconda3/lib/5.26.2/x86_64-linux-thread-multi
/data1/home/Chennan01/miniconda3/lib/5.26.2PATH :是環境變量,它告訴系統在哪裏查找可執行文件。
@INC :是Perl的全局數組,包含了Perl查找模塊的目錄列表。
Shebang腳本解釋器:(#!)是腳本文件的第一行,告訴系統用哪個解釋器執行此腳本。讀取第一行後會使用該目錄下的程序運行腳本。
原因:PATH 讓系統找到個人miniconda的hisat2,Shebang 讓腳本使用個人miniconda的perl,但@INC 包含了系統anaconda的模塊路徑。而miniconda和anaconda兩者的perl版本不一致
[Chennan01@login software]$ hisat2
Can't load '/share/apps/anaconda3/lib/5.26.2/x86_64-linux-thread-multi/auto/Fcntl/Fcntl.so' for module Fcntl: /share/apps/anaconda3/lib/5.26.2/x86_64-linux-thread-multi/auto/Fcntl/Fcntl.so: undefined symbol: Perl_newSV_type at /share/apps/anaconda3/lib/5.26.2/XSLoader.pm line 96.
at /share/apps/anaconda3/lib/5.26.2/x86_64-linux-thread-multi/Fcntl.pm line 66.//在 require 中編譯失敗
Compilation failed in require at /share/apps/anaconda3/lib/5.26.2/x86_64-linux-thread-multi/POSIX.pm line 17.//BEGIN 失敗——編譯在...處中止
BEGIN failed--compilation aborted at /share/apps/anaconda3/lib/5.26.2/x86_64-linux-thread-multi/POSIX.pm line 17.
Compilation failed in require at /data1/home/Chennan01/miniconda3/bin/hisat2 line 35.
BEGIN failed--compilation aborted at /data1/home/Chennan01/miniconda3/bin/hisat2 line 35.1. 加載Fcntl.so時,發現需要解析符號"Perl_newSV_type"
2. 在當前進程的全局符號表中查找:
- 主程序(perl解釋器)的符號表
- 已加載的其他共享庫符號表
3. 如果找不到,記錄為"未定義符號"
4. 運行時嘗試調用該符號時,觸發"SIGSEGV"或"undefined symbol"錯誤
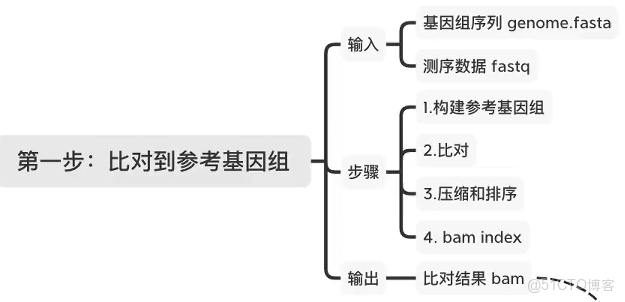
1. 比對
比對到參考基因組
(1)構建參考基因組
# step1:構建參考基因組
less *gz >genome.fasta //將各個染色體序列合併為一個基因組文件
hisat2-build /data1/home/Chennan01/RNA-Seq/data/ref/genome.fa /data1/home/Chennan01/RNA-Seq/data/ref/genome &>hisat2-build.log
# pbs腳本
#!/bin/bash
#PBS -q large_q
#PBS -l nodes=1:ppn=2
#PBS -l mem=8gb
cd /data1/home/Chennan01/RNA-Seq/1.mapping
hisat2-build /data1/home/Chennan01/RNA-Seq/data/ref/genome.fasta /data1/home/Chennan01/RNA-Seq/data/ref/genome &>hisat2-build.log實際使用資源:內存4g左右,單核即可。運行時間:00:13:33
(2)比對
# 輸入文件:-x、-U(或-1、-2)
# 輸出文件:-S
hisat2 --new-summary -p 10 -x ../ref/genome -U ../data/BLO_S1_LD1.fq.gz -S BLO_S1_LD1.sam --rna-strandness R 1>BLO_S1_LD1.log 2>&1
//--new-summary:輸出報告使用新版格式
//-p:線程數
//-x:基因組路徑
//-U:單末端測序,後接文件路徑。雙末端測序則用-1、-2。可為.gz文件
//-S:輸出文件路徑
//--rna-strandnes:鏈特異性,只比對一次。不加這個參數會正向比對一次、反向比對一次。不明確是否特異時則不加。單末端測序接R,雙末端測序接RF
Usage:
hisat2 [options]* -x <ht2-idx> {-1 <m1> -2 <m2> | -U <r>} [-S <sam>]
<ht2-idx> Index filename prefix (minus trailing .X.ht2).
<m1> Files with #1 mates, paired with files in <m2>.
Could be gzip'ed (extension: .gz) or bzip2'ed (extension: .bz2).
<m2> Files with #2 mates, paired with files in <m1>.
Could be gzip'ed (extension: .gz) or bzip2'ed (extension: .bz2).
<r> Files with unpaired reads.
Could be gzip'ed (extension: .gz) or bzip2'ed (extension: .bz2).
<sam> File for SAM output (default: stdout)
<m1>, <m2>, <r> can be comma-separated lists (no whitespace) and can be
specified many times. E.g. '-U file1.fq,file2.fq -U file3.fq'.生成腳本
hisat2 --new-summary -p 10 -x ../ref/genome -U ../data/BLO_S1_LD1.fq.gz -S BLO_S1_LD1.sam --rna-strandness R &>BLO_S1_LD1.log
awk '{print "hisat2 --new-summary -p 10 -x /data1/home/Chennan01/RNA-Seq/data/ref/genome -U "$5" -S "$3".sam --rna-strandness R &>"$3".log"}' /data1/home/Chennan01/RNA-Seq/data/sample.txt >step2_hisat2.pbs
# pbs腳本前添加
#!/bin/bash
#PBS -q large_q
#PBS -l nodes=1:ppn=10
#PBS -l mem=32gb
cd /data1/home/Chennan01/RNA-Seq/1.mapping實際使用資源:3g內存,非線性。一個樣本比對用時1分鐘左右
比對結果
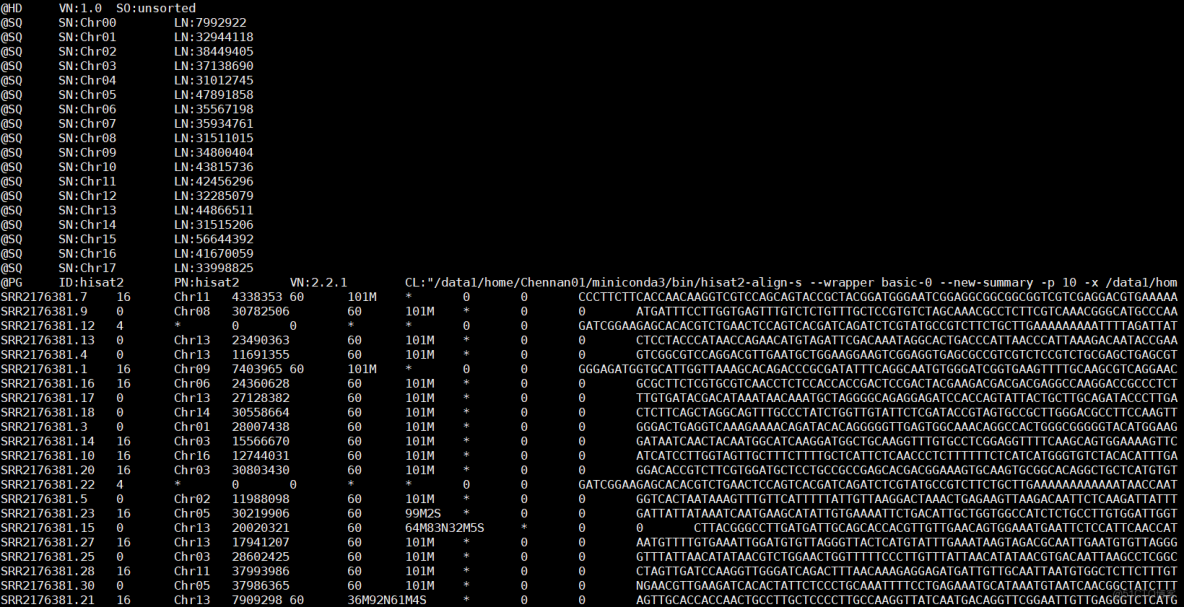
[Chennan01@login 1.mapping]$ cat BL0_S1_LD1.log
HISAT2 summary stats:
Total reads: 15774283
Aligned 0 time: 3725609 (23.62%)
Aligned 1 time: 11158094 (70.74%)
Aligned >1 times: 890580 (5.65%)
Overall alignment rate: 76.38%合併比對信息
for i in *log; do echo "${i%.log}:" >>hisat2.log; cat $i >>hisat2.log; echo -e "\t" >>hisat2.log; done(3)壓縮和排序
將sam格式轉換為bam格式。(sam未排序、太佔內存)
samtools sort -o BLO_S1_LD1.bam BLO_S1_LD1.sam
# 生成腳本
awk '{print "samtools sort -o "$3".bam "$3".sam"}'/data1/home/Chennan01/RNA-Seq/data/sample.txt
# 或者
#!/bin/bash
#PBS -q large_q
#PBS -l nodes=1:ppn=10
#PBS -l mem=32gb
cd /data1/home/Chennan01/RNA-Seq/rnaseq-apple-training/RNASeq-ref/1.Mapping
for i in *.sam; do
samtools sort -o "${i%.sam}.bam" "$i" &>"${i%.sam}.samtobam.log"
//$var:變量引用。${var}:明確變量邊界。${var%pattern}:刪除最小後綴。${var%%pattern}:刪除最長後綴。
//"":防止將帶空格的變量分割成幾個,這裏可不加
done
# 或不加“;”
for i in *sam
do
samtools sort -o "${i%.sam}.bam" "$i"
donebash中引號的作用
# 雙引號: 允許變量擴展
name="John Doe"
echo "Hello, $name" // Hello, John Doe
# 單引號: 禁止所有擴展
echo 'Hello, $name' // Hello, $name
# 無引號: 會進行單詞分割和通配符擴展。
#(1)Shell 默認用空格、製表符、換行符(IFS)來拆分變量值。
files=*.txt
echo $files // 展開為所有.txt文件列表
name=John Doe
echo "Hello, $name" // Hello, John Hello, Doe
#(2)Shell 在解析命令前會自動將通配符(*、?、[])替換為匹配的文件名列表
files="*.sam"
echo $files // *.sam。雙引號內不支持通配符擴展bash中$的用法
# 刪除後綴
fi="my.file.tar.gz"
echo ${fi%.*} # 輸出: my.file.tar。刪除.gz
echo ${fi%%.*} # 輸出: my。刪除.file.tar.gz實際使用資源:5G內存。一個樣本轉換用時約4分鐘
(4)bam index【可選】
生成bai文件,供IGV使用
step4_bamindex.pbs
samtools index BLO_S1_LD1.bam
# pbs腳本
#!/bin/bash
#PBS -q large_q
#PBS -l nodes=1:ppn=10
#PBS -l mem=32gb
cd /data1/home/Chennan01/RNA-Seq/1.mapping //不切換目錄,日誌文件輸出默認在home目錄
for i in *bam; do
echo -e "\n$i:" &>>bam_index.log; //echo默認不解釋轉義字符,加-e才解析。\n換行
samtools index $i &>>bam_index.log;
done問題:是否需要組裝轉錄本再進行比對?不需要
1)在模式物種上,基因註釋到達isoform水平:轉錄組比對時,有的reads沒有對應外顯子
- 該read屬於未標註基因的外顯子:發現新基因
- 該read屬於已標註基因的外顯子,但外顯子未標註:優化基因結構
- 發現前人未報道過的新的轉錄本,如內含子保留。【轉錄本組裝】
2)非模式物種上,基因註釋到達基因水平,未研究mRNA怎麼剪接
3)無參考基因組,拼接轉錄本後再進行比對錶達定量。
- trinity
- stringtie:lncRNA在現有註釋中幾乎都不完整,每個物種每個個體lncRNA都不一樣
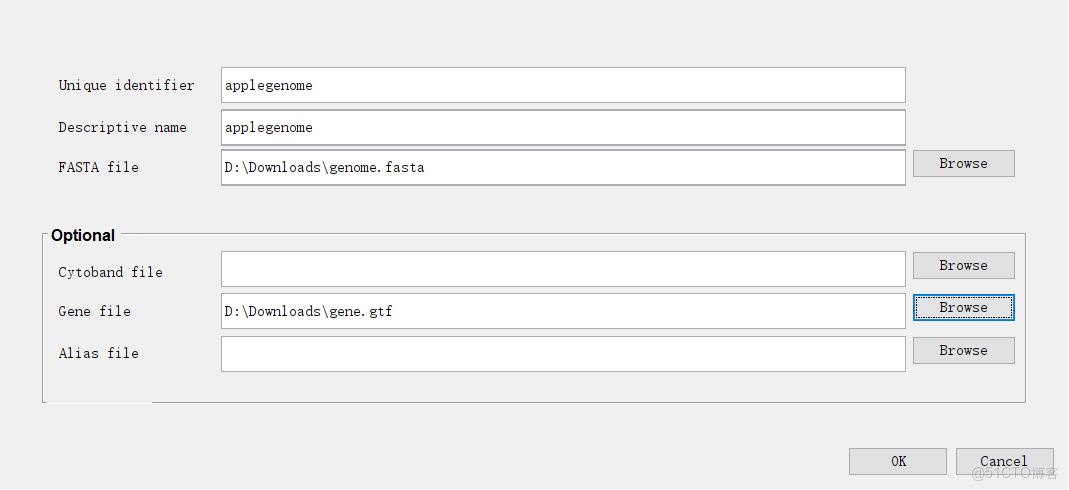
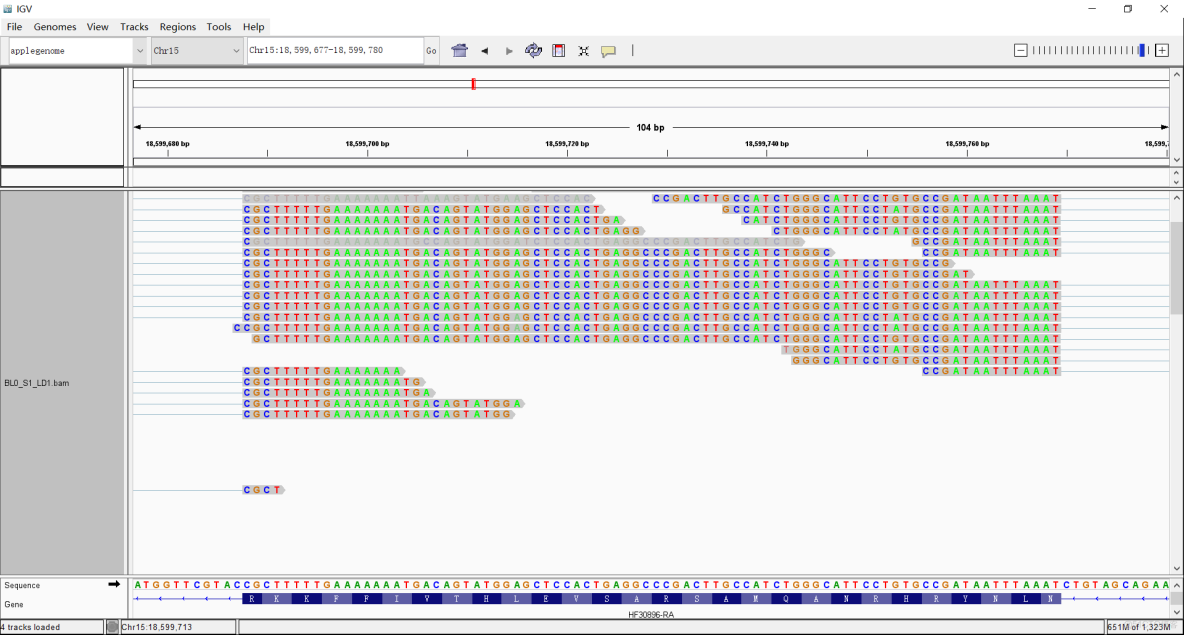
(5)比對結果可視化:igv
1)導入基因組genome.fasta
2)導入基因註釋文件gene.gtf
3)加載比對結果BL0_S1_LD1.bam,要求有索引文件BL0_S1_LD1.bam.bai。show all bases
比對結果只有一個方向對齊則是鏈特異性文庫,兩個則為非特異性文庫