挖呀挖!AutoDock 分子對接極簡實踐方法👋
哈哈哈 開心😆
在小小的花園裏面挖呀挖呀挖 🌱
種下一顆小小的種子🫘
開出一朵小小的花🌸
在大大的花園裏面挖呀挖呀挖🌱
種下一顆大大的種子🫘
開出一朵大大的花🌸
挖呀挖呀挖🌱!更多的是一顆擁抱童真的心😁,在科研和生活之間找到平衡感,並保留一份屬於自己的空間。
分子對接是一種廣泛應用於藥物發現的基於計算機結構的方法。對接能夠識別具有治療意義的新型化合物,在分子水平上預測配體-靶標相互作用,或描繪結構-活性關係 (SAR),而無需先驗地瞭解其他靶標調節劑的化學結構。
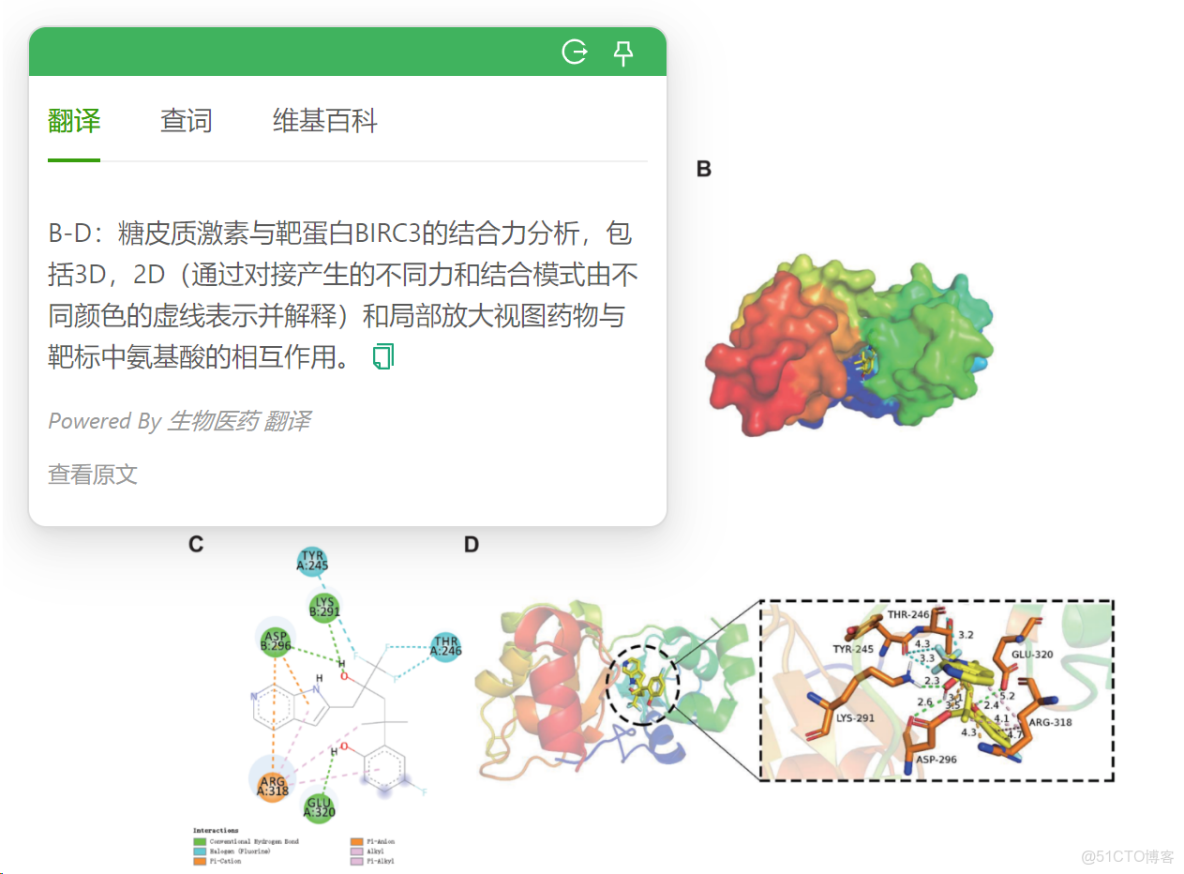
今天大軒來和大家一起來複現一篇發表在International Journal of Molecular Sciences雜誌(IF=6.208,JCR1區)一篇SCI論文中的分子對接模式圖。
Binding efficacy of key genes to small molecule drugs
Part1復現思路
Part2視頻教程
《挖呀挖!AutoDock 分子對接極簡實踐方法👋》視頻教程在B站空間“大軒的成長花園”發佈哦!
Part3實踐方法
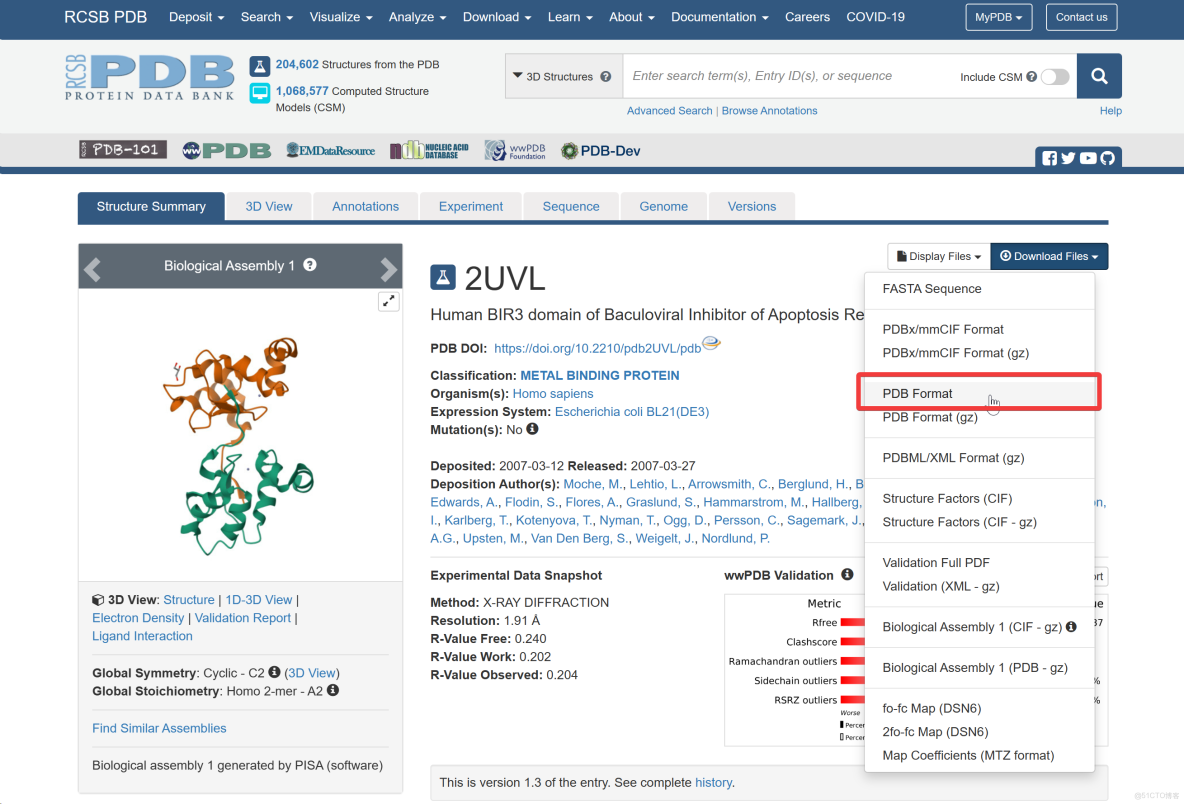
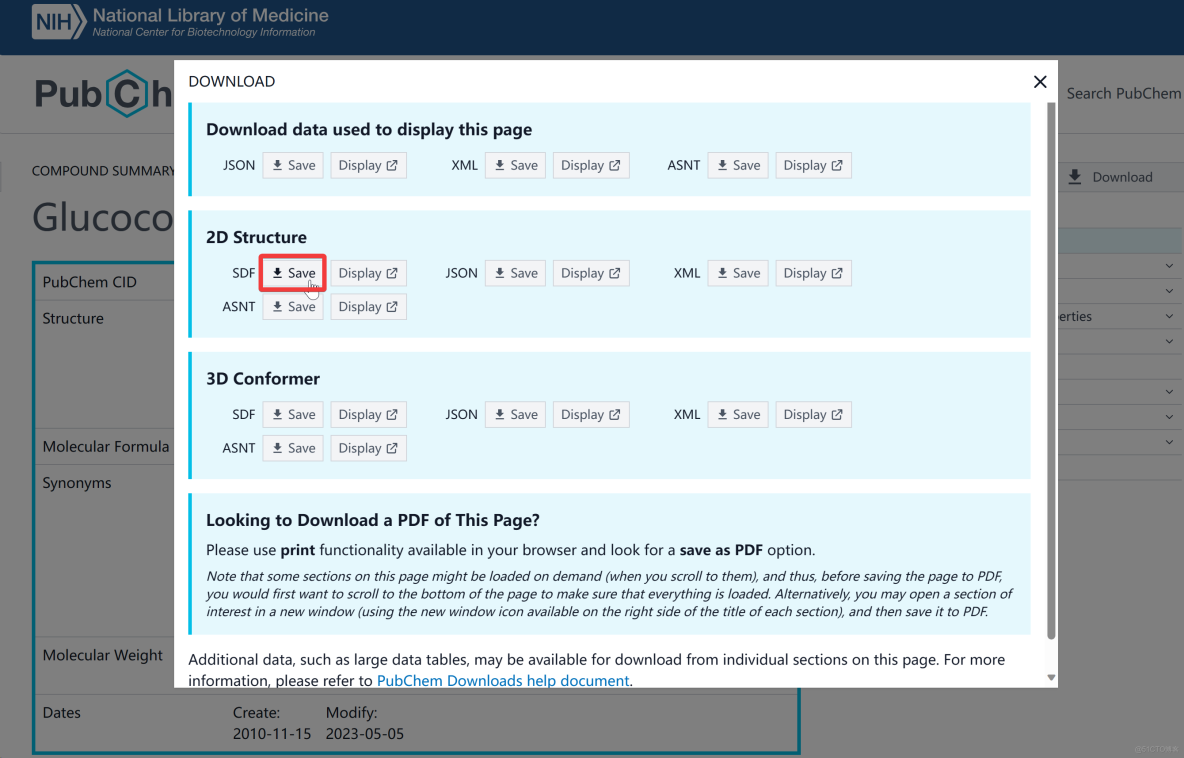
1下載蛋白與化合物結構文件
BIRC3
- 作為受體,
Glucocorticoid
- 作為配體。
-
- 從
RCSB PDB數據庫(http://www.rcsb.org/)中找到受體蛋白BIRC3的3D結構模式,保存為“PDB”格式文件。 -
- 從
PubChem數據庫下載Glucocorticoid的2D結構。
2受體與配體的結構預處理
- 我們將使用
Chem3D
- 、
Pymol
- 、
AutoDock Vina
- 軟件來對已經下載好的結構文件進行優化和預處理。
-
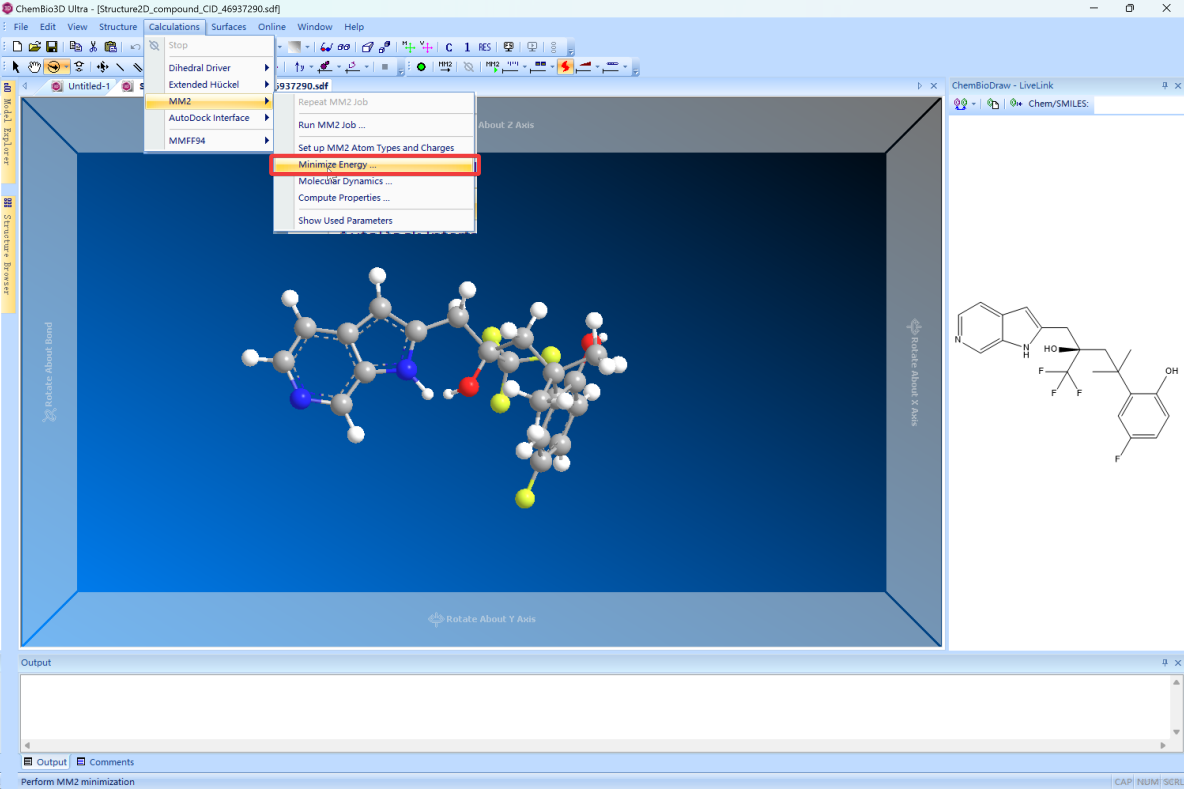
- 針對
Glucocorticoid配體文件,導入Chem 3D軟件,進行能量最小化,輸出為“MOL2”格式以保存其3D結構,並導入AutoDock Tools 1.5.6軟件再輸出為“PDBQT”格式文件。 -
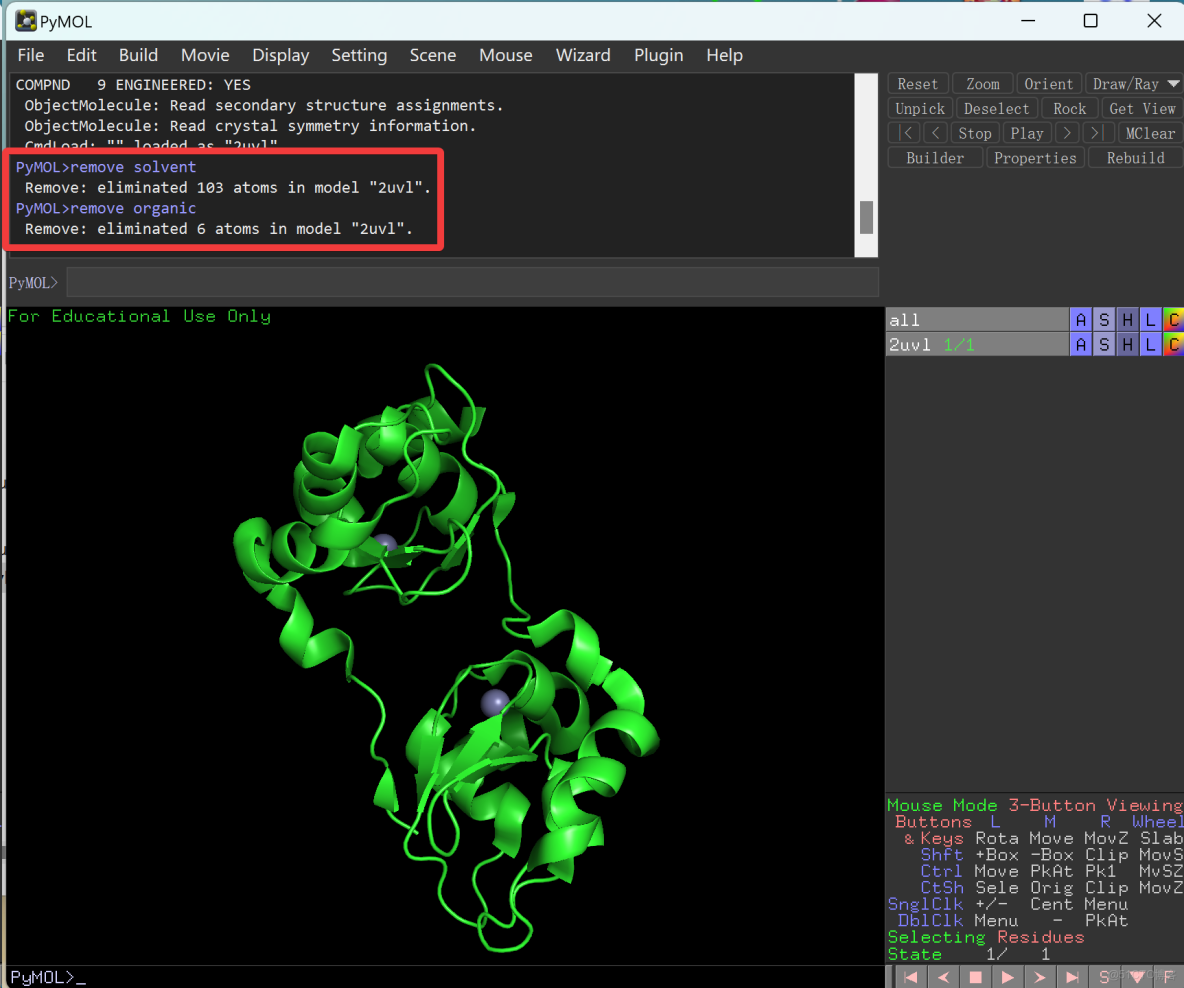
- 針對
BIRC3受體文件,利用PyMOL 2.4.0軟件去除水分子和蛋白質受體中的原有配體,另存為pdb格式。 -
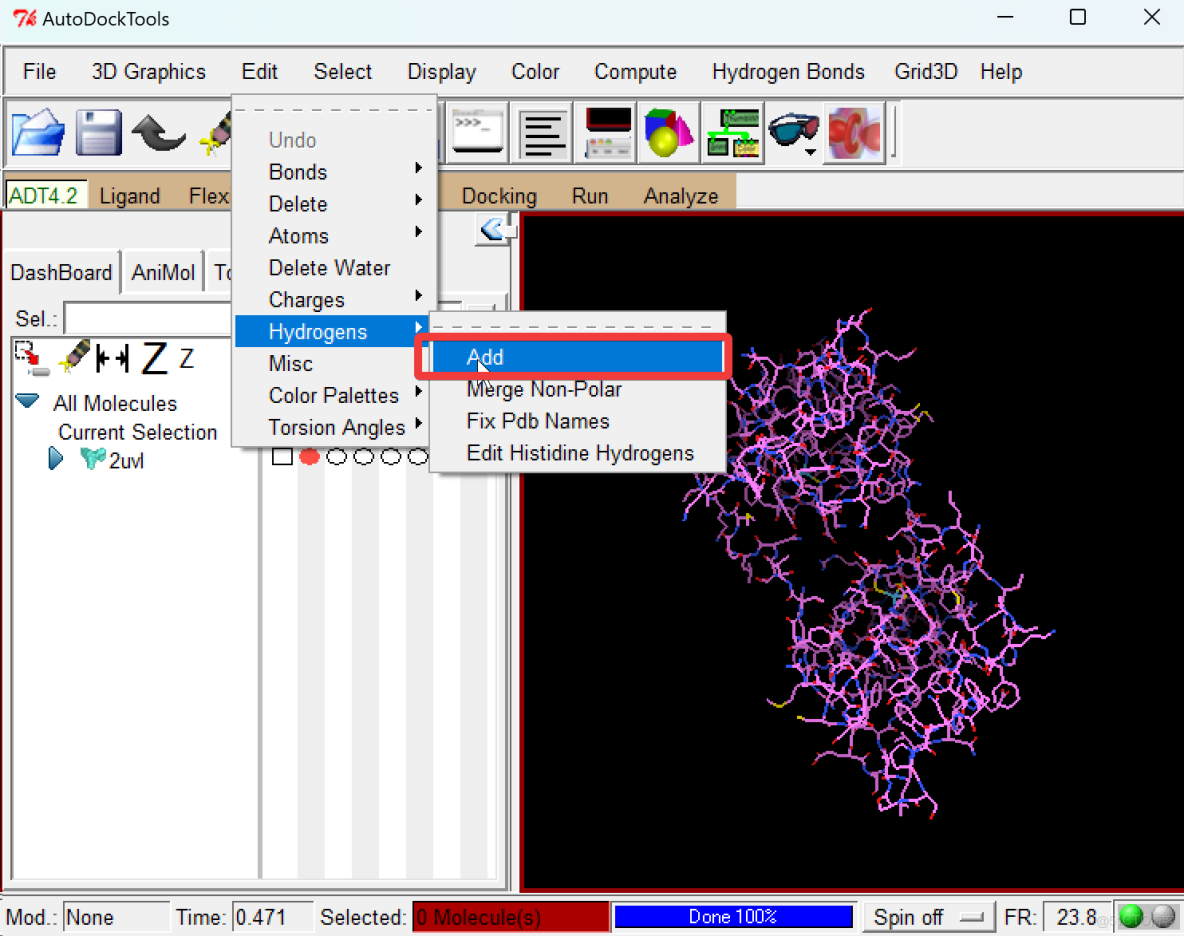
- 使用
AutoDock Tools 1.5.6軟件對受體蛋白加氫處理,並輸出為“PDBQT”格式文件。
Pymol命令行快速操作
remove solvent remove organic
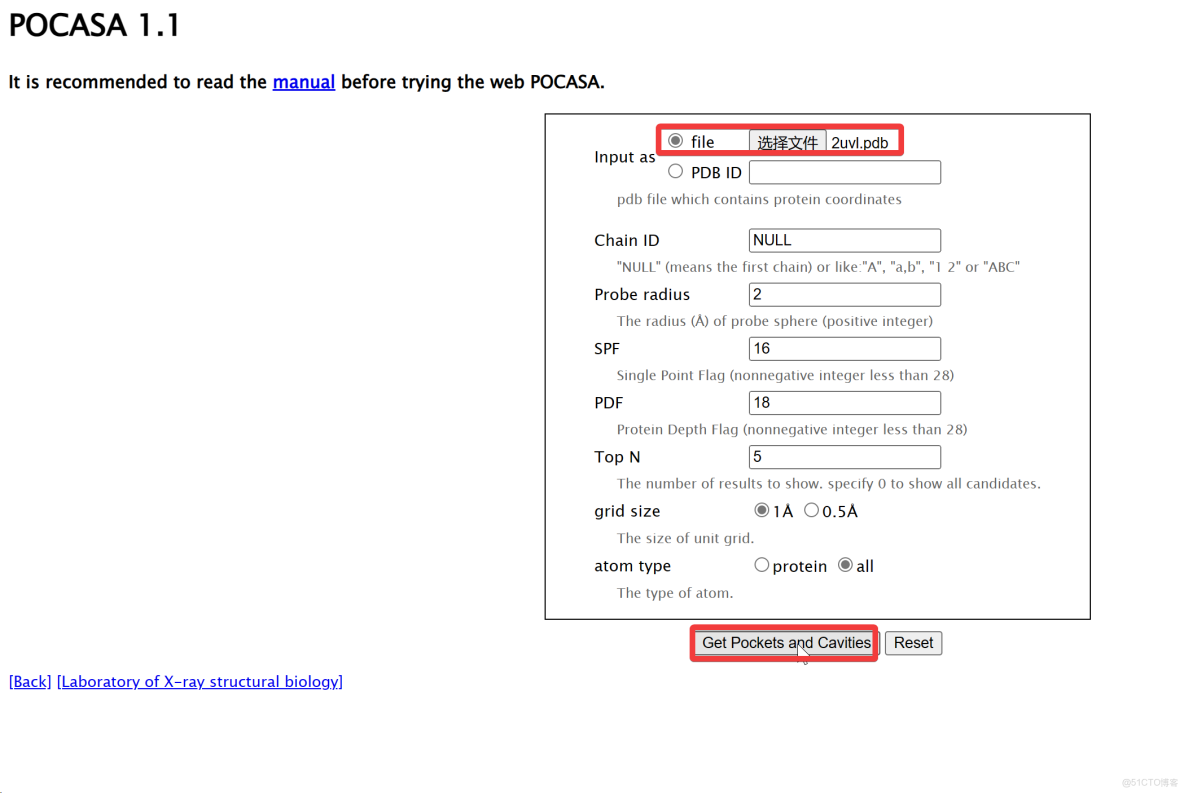
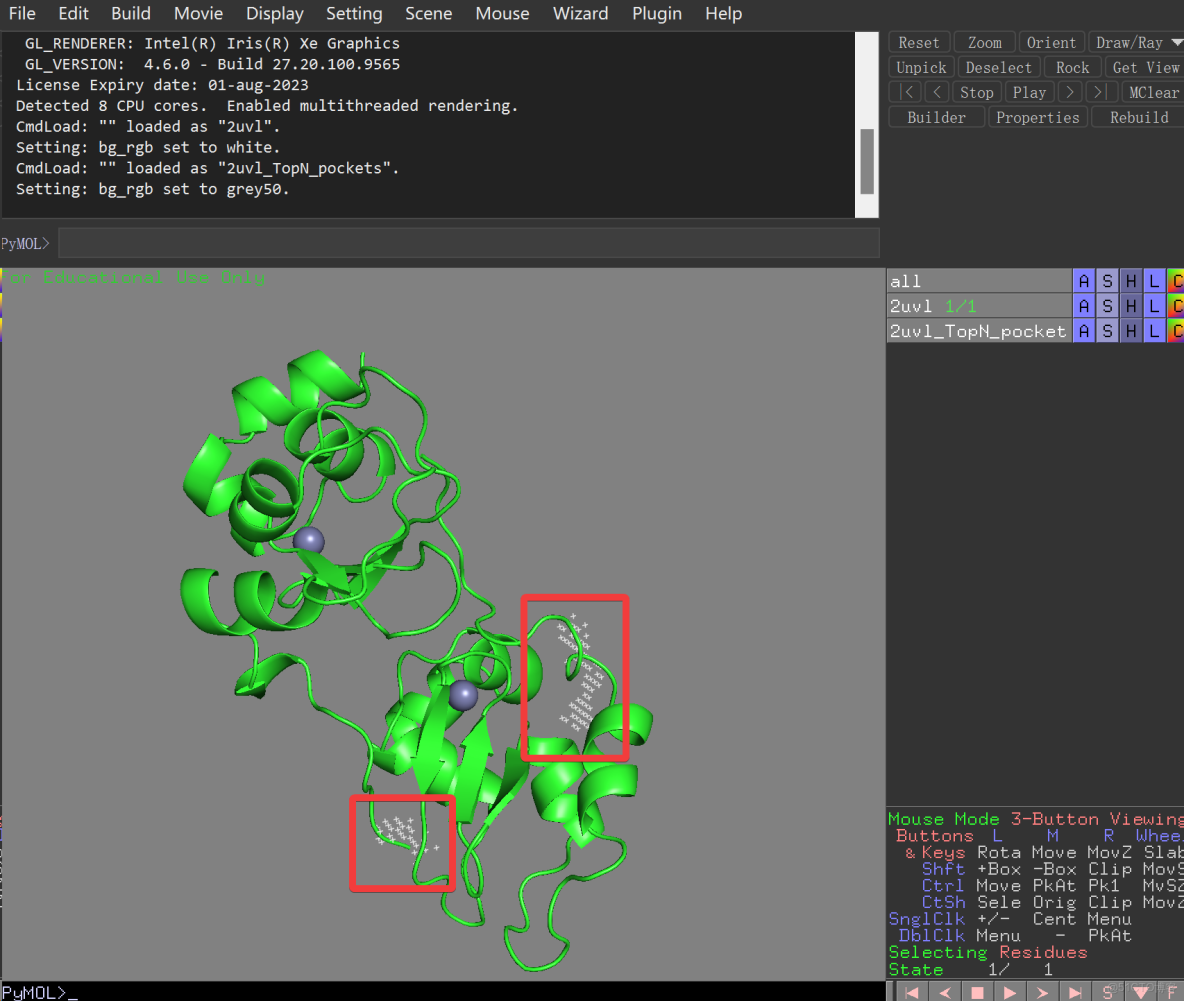
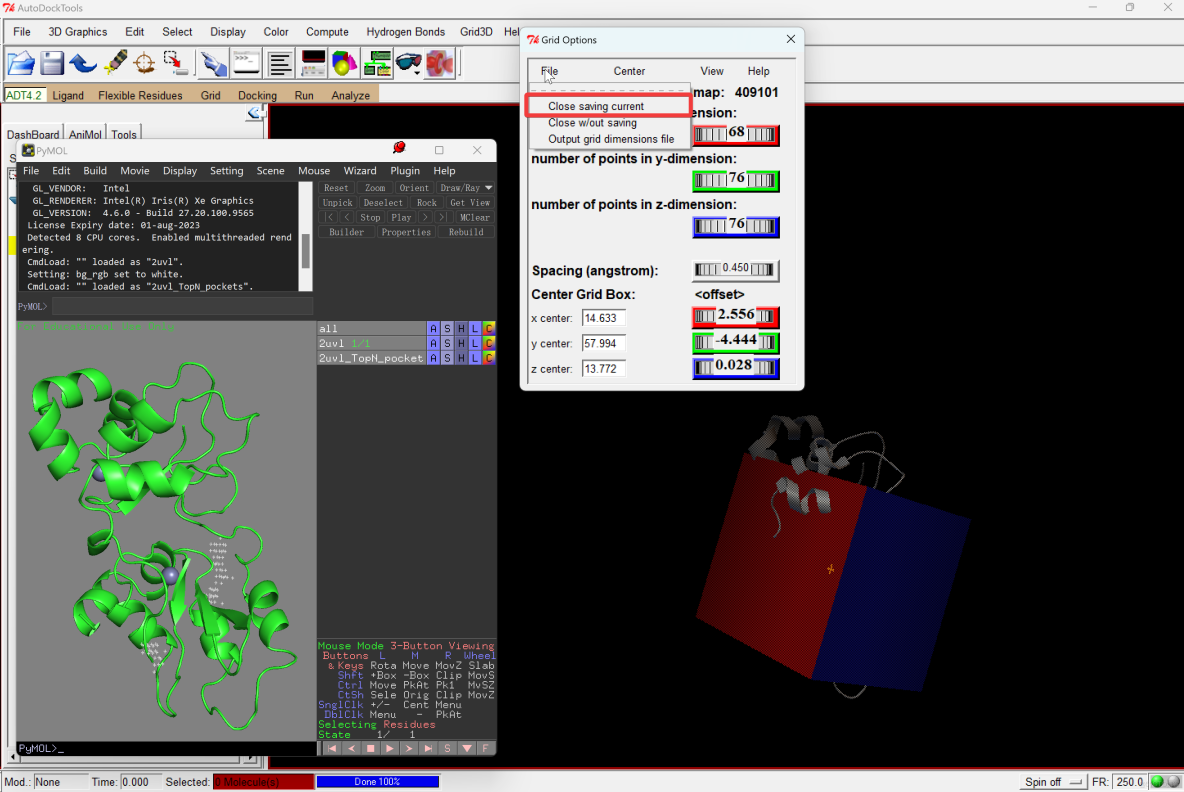
3尋找活性口袋並設置參數
POCASA網站
- 來預測蛋白質潛在的活性口袋
AutoDock Tools 1.5.6
- 軟件設置對接盒子的參數
4AutoDock分子對接
AutoDock Tools 1.5.6
- 軟件進行分子對接。
5對接結果可視化
PyMOL
- 和
Discovery Studio進行可視化分析。詳情見視頻吧!
Part5共勉
下面這句話是大軒在學習分子對接過程中,被軟件各種報錯折磨時(這也是大軒建立知識星球的原因),在ResearchGate上面一位善良的大佬鼓勵大軒的一句話,大軒觸動很深,和大家共勉:
Part6演示文件與安裝包
大軒的AutoDock
- ,我會把全部演示文件與分子對接軟件安裝包發給你,幫助你來複現實操以快速掌握分子對接技術。
因水平有限,有錯誤的地方,歡迎批評指正!
Part7參考資料📚
- Molecular Docking: Shifting Paradigms in Drug Discovery(PMID:31487867)
- Single-Cell Sequencing-Based Validation of T Cell-Associated Diagnostic Model Genes and Drug Response in Crohn's Disease(PMID:37047025)
- Combining machine learning systems and multiple docking simulation packages to improve docking prediction reliability for network pharmacology(PMID: 24391846)
- 利用autudock vina將1000個配體與蛋白質進行分子對接
- POCASA網站
- 如何確定對接口袋?
- ResearchGate
大軒邀請你來玩FlowUs
FlowUs作為新一代知識管理與協作平台,以雲端筆記為載體,配合在線文檔、知識庫、文件夾等多形態功能,為個人和團隊提供數字信息管理與協同的一站式工作中心。