文章和代碼已經歸檔至【Github倉庫:https://github.com/timerring/dive-into-AI 】或者公眾號【AIShareLab】回覆 R語言 也可獲取。
缺失值處理
在實際的數據分析中,缺失數據是常常遇到的。缺失值(missing values)通常是由於沒有收集到數據或者沒有錄入數據。
例如,年齡的缺失可能是由於某人沒有提供他(她)的年齡。大部分統計分析方法都假定處理的是完整的數據集。因此,除了一些專業化的書籍,大多數統計學教科書很少涉及這一問題。實際上,在進行正式的分析之前,我們需要在數據準備階段檢查數據集是否存在缺失值,並通過一些方法彌補因缺失值所造成的損失。
1. 識別缺失值
在 R 中,缺失值用 NA 表示,是“Not Available”的縮寫。函數 is.na( ) 可以用於識別缺失值,其返回結果是邏輯值 TRUE 或 FALSE。
height <- c(100, 150, NA, 160)
height
# 100 150 <NA> 160
is.na(height)
# FALSE FALSE TRUE FALSE如果數據很少,缺失值的個數直接可以數出來,比如上面的變量 height 只有一個缺失值。但是如果數據量很大,就需要藉助函數 table( ) 了。
table(is.na(height))
# FALSE TRUE
# 3 1 需要注意的是,任何包含 NA 的計算結果都是 NA。例如:
mean(height)
# <NA>想要得到所有可參與計算的元素的平均值,應該先將 NA 從向量中移除。
mean(height, na.rm = TRUE)
# 136.666666666667參數 na.rm 表示移除缺失值,其意義與用函數 na.omit( ) 把缺失值省略是一樣的。
mean(na.omit(height))注意,這裏 na.omit( ) 是一個獨立的函數,它能忽略輸入對象中的缺失值,而 na.rm 只是計算描述性統計量的函數裏的一個內部參數。
函數 summary( ) 在計算向量的統計量時會自動忽略缺失值,它會給出向量中缺失值的個數。例如:
summary(height)
# Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
# 100.0 125.0 150.0 136.7 155.0 160.0 1 2. 探索數據框裏的缺失值
在決定如何處理缺失值之前,瞭解哪些變量有缺失值、數目有多少、是什麼組合形式等是非常有意義的。下面用一個示例介紹探索缺失值模式的方法。
datasets 包裏的數據集 iris 也稱鳶尾花數據,它包含 150 個鳶尾花樣品,分為 3 個品種(Species),每個品種各有 50 個樣品。每個樣品又包含 4 個屬性,即花萼長度(Sepal.Length)、花萼寬度(Sepal.Width)、花瓣長度(Petal.Length)和花瓣寬度(Petal.Width)。該數據集不含缺失值。為了説明缺失值的處理方法,首先人為地生成一些缺失數據,以探索缺失值的模式和檢驗補全的效果。
missForest 包裏的函數 prodNA( ) 可以隨機生成缺失值,使用此函數前需要安裝和加載 missForest 包。
options(warn=-1)
library(missForest)
data(iris)
# 為了使結果具有可重複性,我們用函數 `set.seed( )` 設置了生成隨機數的種子。
set.seed(1234)
# 函數 prodNA( ) 默認生成數據數目 10% 的缺失值,我們可以通過改變參數 noNA 的值以生成不同數目的缺失值。
iris.miss <- prodNA(iris)
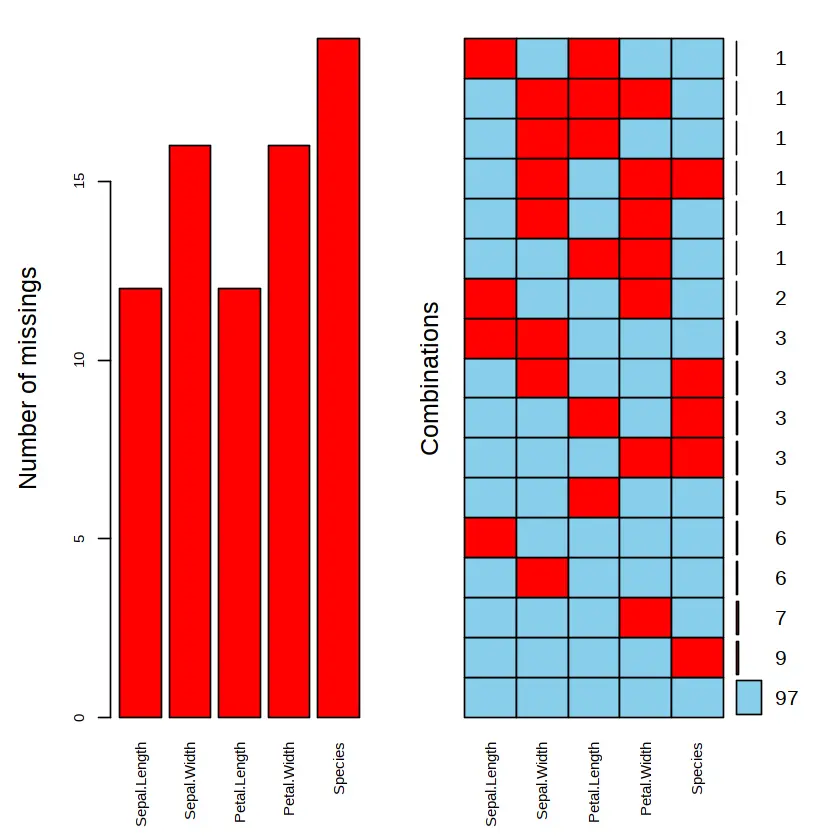
summary(iris.miss)從函數 summary( ) 的輸出中可以看到每個變量裏缺失值的數目。要了解數據集裏缺失值的模式,用圖形展示是一個好辦法。VIM 包提供了大量可視化缺失值的函數,其中函數 aggr( ) 不僅展示每個變量裏缺失值的個數(或比例),還展示多個變量組合下缺失值的個數(或比例)。例如:
library(VIM)
aggr(iris.miss, prop = FALSE, numbers = TRUE, cex.axis = 0.7)在上圖中,第一幅圖是用條形圖展示了每個變量缺失值的個數,這與上面函數 summary( ) 的輸出結果是一致的;第二幅圖展示了數據框中 5 個變量不同組合下缺失值的個數,其中紅色方塊代表缺失值,最右邊的數字代表個數。從最下面看起,共有 97 個鳶尾花樣品沒有缺失值,有 9 個鳶尾花樣品知道它們的 4 個屬性但不知道品種。
3. 填充缺失值
一般來説,處理缺失值可以採用下面 3 種方法:
- 刪除,刪除帶有缺失值的變量或記錄;
- 替換,用均值、中位數、眾數或其他值替代缺失值;
- 補全,基於統計模型推測和補充缺失值。
上述方法都是在不得已時使用,無論哪種方法都不能完全彌補數據缺失帶來的信息損失。因此,在數據收集階段必須儘量避免數據的缺失。
3.1 刪除缺失值:na.omit( )、complete.cases( )
如果缺失值的數量很小,刪除後對分析結果影響不大,我們可以使用前面提到的函數 na.omit( ) 刪除數據框中的缺失值。例如:
iris.sub <- na.omit(iris.miss)
nrow(iris.sub)刪除缺失值後的數據框 iris.sub 只包含 97 條完整記錄。此外,函數 complete.cases( ) 可以用來識別矩陣或數據框中沒有缺失值的行,它的返回值是 TRUE 或 FALSE。如果某一行有完整的數據,返回 TRUE;如果某一行至少包含一個缺失值,則返回 FALSE。所以,上面的命令等價於:
iris.sub <- iris.miss[complete.cases(iris.miss), ]3.2 使用特定數值替換缺失值
如果不想直接刪除缺失值,在某些情況下,還可以嘗試使用特定的數值替換缺失值。
下面以變量 Sepal.Length 為例,用忽略缺失值後的均值替換該變量裏的缺失值。先計算均值:
Sepal.Length.Mean <- mean(iris.miss$Sepal.Length, na.rm = TRUE)
Sepal.Length.Mean
# 5.78695652173913
# 用忽略缺失值後的均值替換該變量裏的缺失值
iris.miss1 <- iris.miss
iris.miss1$Sepal.Length[is.na(iris.miss1$Sepal.Length)] <- Sepal.Length.Mean為檢查補全後的數據與原始數據的差異,我們可以計算偏差:
summary((iris$Sepal.Length - iris.miss1$Sepal.Length)/iris$Sepal.Length)
# Min. 1st Qu. Median Mean 3rd Qu. Max.
# -0.258034 0.000000 0.000000 0.006871 0.000000 0.248447補全的平均偏差不到 1%,但最大偏差大約為 ±25%。
3.3 多重插補
多重插補(multiple imputation)是一種基於重複模擬的處理缺失值的方法,常用於處理比較複雜的缺失值問題。
R 中有多個可以實現缺失值多重插補的包,如 Amelia 包、mice 包和 mi 包等。其中 mice 包使用鏈式方程的多變量補全法,被廣泛運用於數據清洗過程中。
mice 包假設數據是隨機缺失的,並根據變量的類型建立模型得到預測值以代替缺失值。在這些模型裏,常用的有:
- 預測均值匹配(pmm),實質上就是線性迴歸,適用於數值型變量;
- Logistic 迴歸(logreg),適用於二分類變量;
- 多分類 Logistic 迴歸(ployreg),適用於無序多分類變量;
- 比例優勢比模型(polr),適用於有序多分類變量。
接下來,用函數 mice( ) 補全數據框 iris.miss 裏的缺失值。
library(mice)
imputed.data <- mice(iris.miss, seed = 1234)
summary(imputed.data)
# PredictorMatrix:
# Sepal.Length Sepal.Width Petal.Length Petal.Width Species
# Sepal.Length 0 1 1 1 1
# Sepal.Width 1 0 1 1 1
# Petal.Length 1 1 0 1 1
# Petal.Width 1 1 1 0 1
# Species 1 1 1 1 0在上面輸出結果的矩陣 PredictorMatrix 裏,每一行代表含有缺失值的變量名,如果該行對應的某一列元素為 1,代表該列變量被用於建模預測。
從上面的輸出結果中可以看出,對於每一個變量,其餘變量都被用於它的缺失值預測。函數 mice( ) 的輸出結果是一個列表,其中的對象 imp 也是一個列表,存放的是每個變量缺失值的插補值。例如,使用下面的命令可以得到變量 Sepal.Length 的插補值:
imputed.data$imp$Sepal.Length函數 mice( ) 通過 Gibbs 抽樣完成,默認進行 5 次隨機抽樣,所以一共得到了 5 組插補值。我們可以通過查看上面的輸出結果以檢查插補值是否合理,然後選擇其中的一組來補全。
例如,取 5 組插補值中的第 3 個:
complete.data <- complete(imputed.data, 3)為了檢查缺失值的補全效果,對於數值型變量,我們可以計算插補值與原始變量值的偏差。
以變量 Sepal.Length 為例:
summary((iris$Sepal.Length-complete.data$Sepal.Length)/iris$Sepal.Length)
# Min. 1st Qu. Median Mean 3rd Qu. Max.
# -0.1428571 0.0000000 0.0000000 0.0007643 0.0000000 0.0945946補全的平均偏差不到 0.1%,最大偏差大約為 ±13%。因此,這裏用多重插補法比用均值替換缺失值的方法效果更好。
數據框的最後一個變量 Species 是一個因子,包含 19 個缺失值。為了檢查這種分類變量的缺失值的補全效果,我們可以用函數 table( ) 得到原始變量和插補後變量的列聯表:
table(iris$Species, complete.data$Species)
# setosa versicolor virginica
# setosa 50 0 0
# versicolor 0 50 0
# virginica 0 1 49這種表被稱為 混淆矩陣(confusion matrix),經常用於評價模型預測的準確度。對角線上的數字代表預測值和真實值一致的個數,非對角線上的數字代表預測值和真實值不一致的個數。
從上面的輸出結果可以看出,變量 Species 的 19 個缺失值插補的正確率為 100%。