全文鏈接:https://tecdat.cn/?p=44689
原文出處:拓端數據部落公眾號
關於分析師
San Zhang
在此對San Zhang對本文所作的貢獻表示誠摯感謝,他在北京航空航天大學完成了控制工程專業的碩士學位,專注人工智能領域。擅長Python、Matlab仿真、視覺處理、神經網絡、數據分析 。
San Zhang曾在所有保險領域工作,包括人壽、健康、汽車、商業險和專業賠償,並在南非、摩洛哥、尼日利亞、肯尼亞、博茨瓦納、坦桑尼亞和加納等非洲市場工作。最近的參與包括為一家南非人壽和養老金保險公司提供數字化轉型和商業模式簡化方面的建議。
專題名稱:大語言模型Prompt工程驅動的臨牀科研數據智能分析專題
引言
從數據科學視角來看,臨牀科研的核心價值在於通過數據挖掘與分析轉化為可落地的診療優化方案,但當前臨牀科研領域普遍面臨"技術門檻高、效率低"的行業痛點。臨牀工作者往往具備深厚的醫學專業積累,卻缺乏系統的數據科學訓練,難以熟練運用機器學習工具完成科研數據分析全流程;而傳統數據分析工具如Python、R等,對非專業用户的學習成本過高,嚴重製約了臨牀科研的推進效率。
隨着大語言模型技術的快速發展,其在自然語言理解、代碼生成等領域的能力為臨牀科研數字化轉型提供了新路徑。但通用大語言模型在醫學場景應用中存在明顯短板,如提示詞設計專業性不足、輸出結果缺乏醫學適配性等,這成為制約技術落地的關鍵瓶頸。
本文內容改編自過往客户諮詢項目的技術沉澱並且已通過實際業務校驗,該項目完整代碼與數據已分享至交流社羣。閲讀原文進羣,可與800+行業人士交流成長;還提供人工答疑,拆解核心原理、代碼邏輯與業務適配思路,幫大家既懂 怎麼做,也懂 為什麼這麼做;遇代碼運行問題,更能享24小時調試支持。
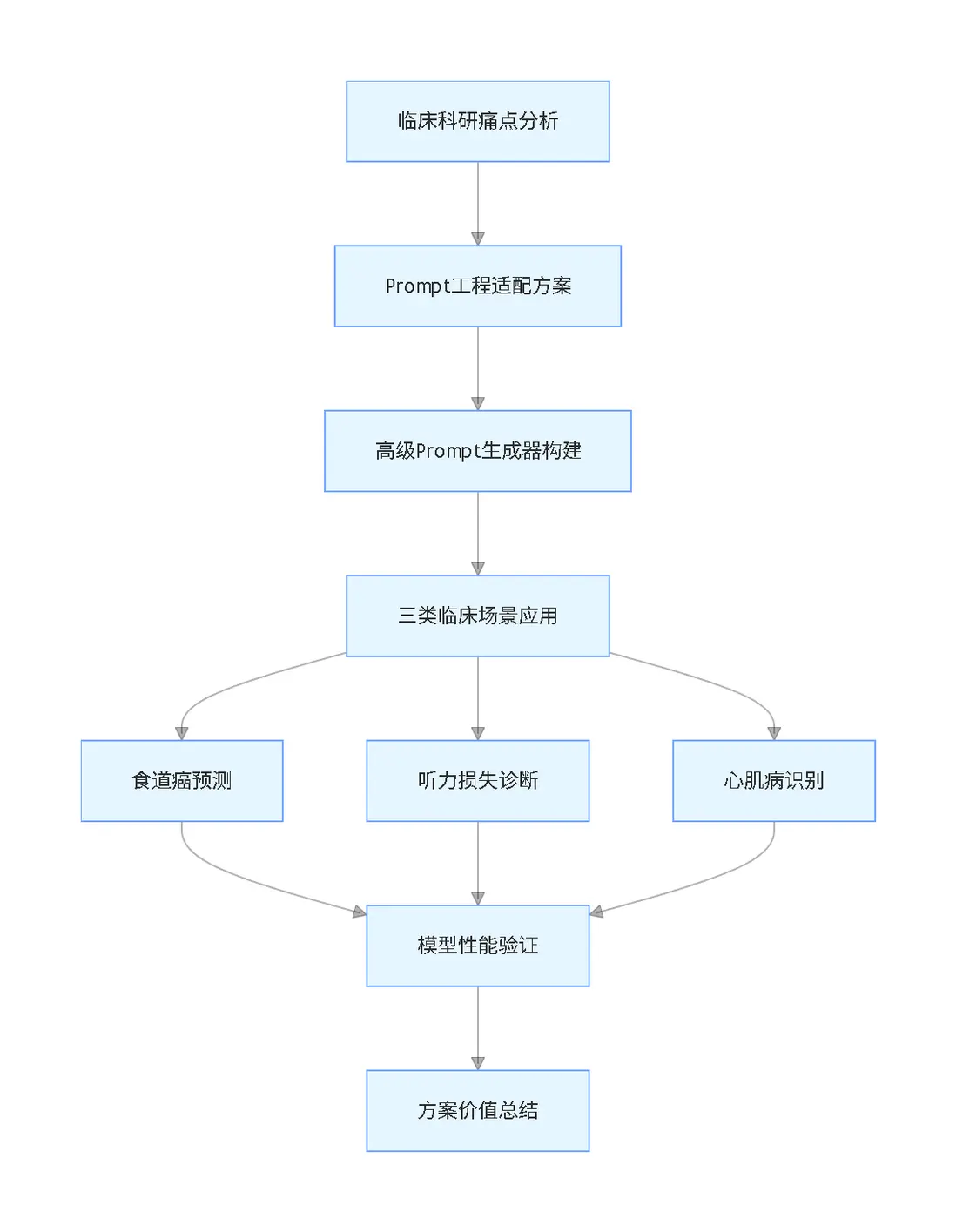
本專題圍繞"大語言模型Prompt工程適配臨牀科研"核心需求,構建高級Prompt生成器,結合邏輯迴歸(LR)、自適應提升(AdaBoost)、輕量梯度提升機(LightGBM)、極端梯度提升(XGBoost)、隨機森林(RF)、支持向量機(SVM)、決策樹(DT)、k-最近鄰(KNN)、多層感知器(MLP)等多種數據分析模型,針對食道癌預測、遺傳性聽力損失診斷、野生型甲狀腺素蛋白澱粉樣變性心肌病識別三類典型臨牀科研場景開展實踐驗證,系統闡述技術方案的設計思路、實現流程與應用效果,為臨牀工作者提供低門檻、高適配的科研數據分析解決方案。
文章脈絡流程圖
項目文件目錄截圖
課題研究背景和意義
醫學研究是推動臨牀實踐進步的核心動力,也是提升患者診療水平的關鍵環節。但在當前臨牀科研實踐中,臨牀工作者面臨諸多現實挑戰:臨牀科研涵蓋研究設計、數據採集、統計分析、模型構建、結果解釋等多個環節,既要求具備醫學專業知識,還需掌握統計學基礎和編程能力。而臨牀工作者大多缺乏系統的數據科學訓練,尤其在機器學習建模、大數據分析等領域存在明顯知識斷層。
現有數據分析工具如Python、R、SPSS等,均要求用户具備一定的編程或軟件操作能力,非專業用户即便完成簡單的數據清洗或迴歸分析,也需投入大量時間學習技術細節,導致科研效率低下。儘管已有部分醫學研究輔助軟件,如文獻管理工具、統計嚮導工具等,但這些工具功能侷限於單一任務,無法為複雜科研流程提供端到端支持。如何將人工智能技術深度融入科研全流程,成為行業亟待解決的難題。
通用型人工智能工具雖具備一定自然語言處理能力,但生成的代碼或分析建議往往缺乏醫學領域針對性,存在術語不準確、方法不專業等問題,難以直接滿足臨牀科研需求。在此背景下,開發一款能夠降低技術門檻、適配醫學領域需求、覆蓋科研全流程的智能輔助系統,已成為提升臨牀科研效率的迫切需求。
近年來,以GPT-4、DeepSeek為代表的大語言模型(LLMs)通過海量文本預訓練和指令微調,展現出強大的自然語言理解、代碼生成和邏輯推理能力,為醫學智能化提供了新可能。在醫學領域,大語言模型的應用潛力主要體現在三個方面:一是文獻解析與知識關聯,可快速解析醫學文獻、臨牀指南和病例報告,生成文獻綜述框架或提取關鍵研究結論;二是研究設計輔助,能基於研究目標推薦合適的研究設計類型和統計分析方法;三是代碼生成與結果解讀,通過自然語言指令自動生成數據預處理、可視化分析或機器學習建模的代碼腳本,還可針對統計分析結果生成符合學術規範的解讀文本。
但現有大語言模型在醫學場景應用中仍存在顯著侷限性:提示詞依賴性強,輸出質量高度依賴輸入提示的設計,非專業用户難以構造精準的醫學任務指令;領域適配不足,通用模型缺乏對醫學專業術語、研究範式的深度理解,易產生邏輯錯誤或方法誤導;交互效率低下,缺乏針對複雜任務的漸進式交互機制,用户需反覆調試提示詞才能獲得理想輸出。這些問題的核心在於如何通過提示詞工程實現大語言模型與醫學場景的高效適配,這也是本研究的核心重點。
本研究聚焦"基於大語言模型的臨牀科研輔助方法"開發,以提示詞設計工程為核心突破點,具備重要的理論和實踐價值。通過構建智能化科研輔助系統,將大語言模型深度嵌入研究設計、數據分析、結果解釋等全流程,實現"自然語言驅動科研"。臨牀工作者可直接簡單描述需求,系統自動生成統計代碼、可視化圖表與方法學建議,顯著降低技術工具使用門檻,推動醫學研究範式革新,解決提示詞設計的醫學適配難題。
相關文章
Python用langchain、OpenAI大語言模型LLM情感分析AAPL股票新聞數據及提示工程優化應用
原文鏈接:https://tecdat.cn/?p=39614
軟件的測試、驗證和評價
測試、驗證和評價方案
本研究通過對比大語言模型生成的研究結果與臨牀已發表論文報道的研究結果,驗證系統的實際應用有效性。選取食道癌預測、變異遺傳性聽力損失診斷、野生型甲狀腺素蛋白澱粉樣變性心肌病識別三類典型臨牀科研場景開展驗證,採用AUROC值、平均精度、F1分數等核心指標進行性能評估。
食道癌的預測
任務説明
食管鱗狀細胞癌和食管胃交界處腺癌預後較差,早期發現是降低死亡率的關鍵。但早期發現依賴上消化道內窺鏡檢查,難以在人羣水平上實施。本場景旨在構建基於機器學習的全自動預測工具,集成微創海綿細胞學測試和流行病學危險因素,用於內窺鏡檢查前的癌症篩查。
測試與驗證
基於已發表研究的食道癌預測任務,提取研究背景、任務説明、數據集信息、模型訓練要求和目標輸出等核心模塊信息,將其輸入"高級Prompt生成器"得到針對性提示詞,再將該提示詞輸入DeepSeek生成對應的Python分析代碼。模型性能主要通過受試者工作特徵曲線下面積(AUROC)和平均精度衡量。
食道癌預測提示詞的生成過程如下:

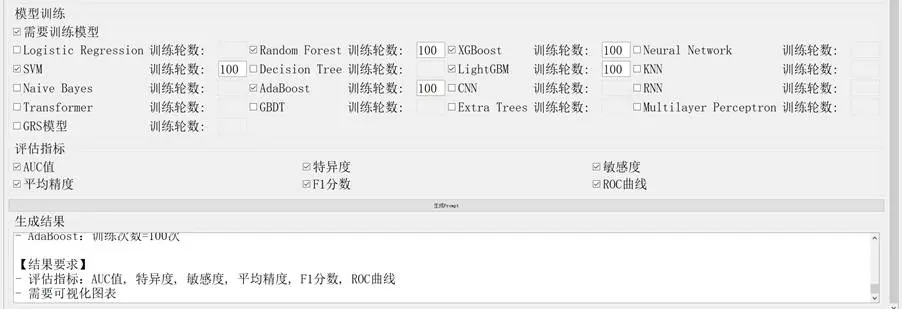
圖2-1 食道癌預測Prompt生成圖
對生成的Python代碼進行文件路徑修改,確保數據集正確讀取。DeepSeek生成的模型在訓練集上的性能表現如下表所示:
表2-1 DeepSeek生成的機器模型性能表現
| 機器學習模型 | AUC值 | 平均精度 |
|---|---|---|
| LightGBM | 0.9479 | 0.4286 |
| AdaBoost | 0.9523 | 0.3825 |
| XGBoost | 0.9403 | 0.4371 |
| SVM | 0.9329 | 0.4128 |
| Random Forest | 0.9448 | 0.3280 |
各模型在測試集中的AUROC值及其特徵曲線如下:
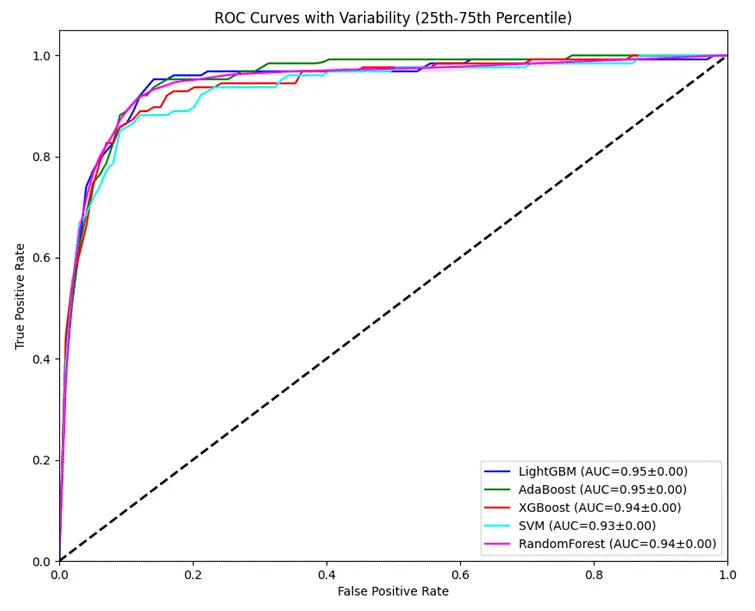
圖2-2 各模型在測試集中的AUROC值及其特徵曲線
已發表研究中作者提供的模型測試結果如下:
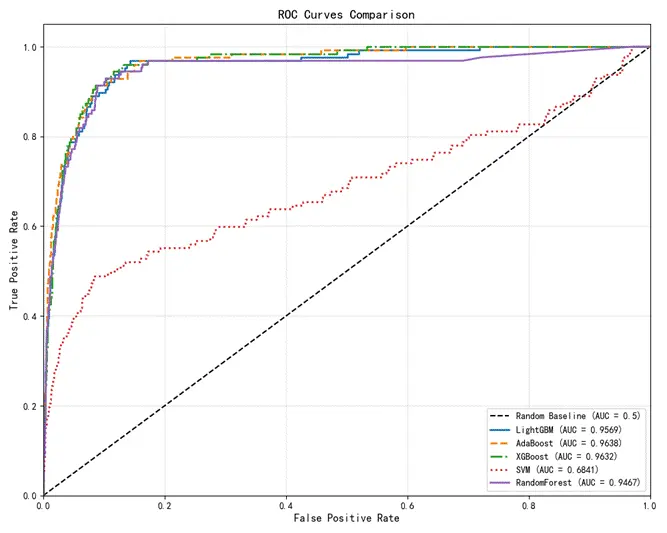
圖2-3 原研究各模型的AUROC值及其特徵曲線
核心代碼實現(修改後)
# 數據準備階段# 讀取訓練集和測試集數據train_data1 = pd.read_csv('./train.csv') # 原始訓練集train_data2 = pd.read_csv('./train_somte.csv') # SMOTE過採樣後的訓練集train_data = pd.concat([train_data1, train_data2], axis=0) # 合併訓練集test_data = pd.read_csv('./test.csv') # 測試集# 定義特徵和目標變量num_features = [f'f{i}' for i in range(1, 106)] # 數值特徵(f1到f105)cat_features = ['Sex', 'Age', 'Urban_rural', 'Smoker', 'SmokingIndex', 'SmokingExtent', 'Drinking', 'DrinkingExtent', 'Flush', 'Pickled', 'HotFood', 'TooothLoss', 'ToothLossNumber', 'GICancer', 'FamilyHistory'] # 分類特徵target_var = 'GroundTruth_bi' # 二分類目標變量# 準備訓練和測試數據集X_train = train_data[num_features + cat_features]y_train = train_data[target_var]X_test = test_data[num_features + cat_features]y_test = test_data[target_var]# 數據預處理階段# 數值特徵處理管道:均值填充缺失值 + 標準化num_transformer = Pipeline(steps=[ ('imputer', SimpleImputer(strategy='mean')), ('scaler', StandardScaler())])# 分類特徵處理管道:眾數填充缺失值 + 獨熱編碼cat_transformer = Pipeline(steps=[ ('imputer', SimpleImputer(strategy='most_frequent')), ('onehot', OneHotEncoder(handle_unknown='ignore'))])# 組合處理器preprocessor = ColumnTransformer( transformers=[ ('num', num_transformer, num_features), ('cat', cat_transformer, cat_features) ])......注:上述代碼省略了10次迭代訓練中的預測及指標計算代碼、ROC曲線繪製的核心代碼,完整代碼可通過交流社羣獲取。
結果對比
已發表研究中表現最佳的模型為LightGBM,其在測試集中的AUROC值為0.960。而DeepSeek生成的模型中,AdaBoost表現最優,AUROC值為0.952,與原始模型性能接近。
對比分析顯示,DeepSeek生成的機器學習模型與重新實現的原始研究模型,AUROC值大致相似(分別為0.952和0.957),無顯著差異,驗證了生成代碼的有效性。
變異遺傳性聽力損失的診斷
任務説明
遺傳性聽力損失是常見的感覺缺陷,嚴重影響患者生活質量。隨着基因測序技術的發展,越來越多的變異被發現,但這些變異的遺傳診斷對專業能力要求極高,人工診斷難度大。本場景旨在構建基於機器學習的遺傳診斷模型,針對GJB2、SLC26A4和MT-RNR1三類遺傳性聽力損失相關變異進行診斷。
測試與驗證
基於已發表研究的遺傳性聽力損失診斷任務,提取核心信息輸入"高級Prompt生成器"得到提示詞,再通過DeepSeek生成Python分析代碼。模型性能通過AUROC值、準確性和F1分數進行評估。
遺傳性聽力損失診斷提示詞的生成過程如下:

圖2-4 HHL診斷Prompt生成圖
對生成的Python代碼進行文件路徑修改後運行,DeepSeek生成的模型性能表現如下表所示:
表2-2 DeepSeek生成的機器模型性能表現
| 機器學習模型 | 準確率 | AUC值 | 平均精度 | F1分數 |
|---|---|---|---|---|
| Decision Tree | 0.728 | 0.660 | 0.848 | 0.837 |
| SVM | 0.764 | 0.700 | 0.855 | 0.866 |
| Random Forest | 0.766 | 0.682 | 0.885 | 0.868 |
| KNN | 0.773 | 0.718 | 0.862 | 0.850 |
| AdaBoost | 0.766 | 0.560 | 0.801 | 0.868 |
| MLP | 0.775 | 0.744 | 0.888 | 0.853 |
各模型在測試集中的AUROC值及其特徵曲線如下:
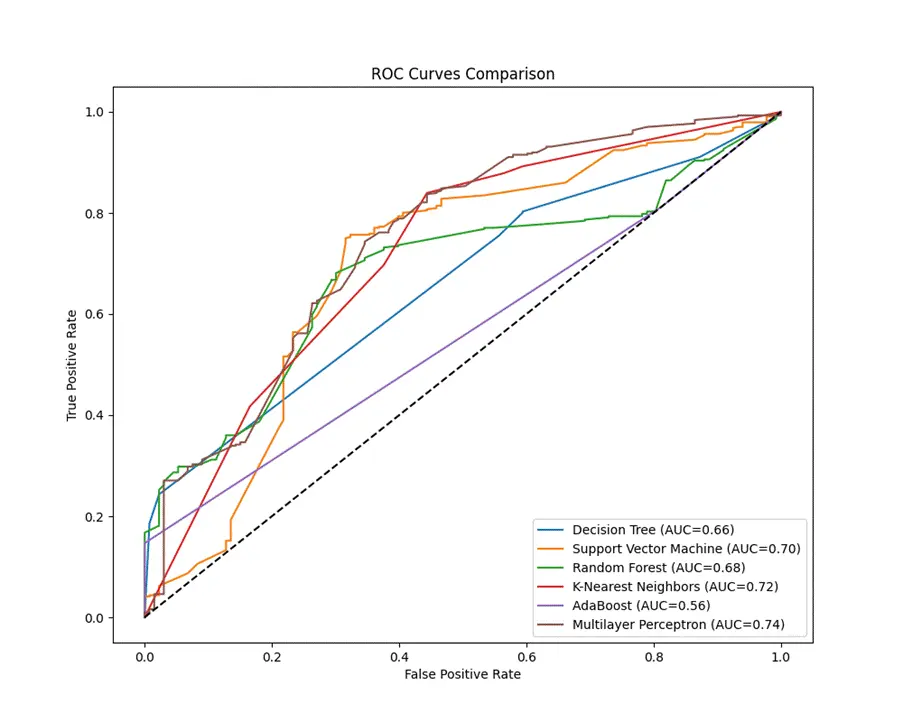
圖2-5 各模型在測試集中的AUROC值及其特徵曲線
核心代碼實現(修改後)
def preprocess_data(train_path, test_path): # 讀取數據 train_df = pd.read_excel(train_path) test_df = pd.read_excel(test_path) # 定義基因相關特徵列(省略具體列定義的詳細代碼) gjb2_cols = ['p.F115C', 'p.M195V', ...] # 省略其他列 mtdna_cols = ['C1494T', 'A1555G', ...] # 省略其他列 slc26a4_cols = ['p.V22E', 'p.V207L', ...] # 省略其他列 all_features = gjb2_cols + mtdna_cols + slc26a4_cols # 列對齊和數據清洗(省略詳細代碼) def align_columns(df): ... train_df = align_columns(train_df) test_df = align_columns(test_df) # 缺失值處理 imputer = SimpleImputer(strategy='most_frequent') X_train = imputer.fit_transform(train_df[final_features]) y_train = train_df['Diagnoses'].values X_test = imputer.transform(test_df[final_features]) y_test = test_df['Diagnoses'].values return X_train, y_train, X_test, y_test# 模型定義函數def get_models(): return { 'Decision Tree': Pipeline([ ('scaler', StandardScaler()), ('clf', DecisionTreeClassifier(max_depth=5, random_state=42)) ]), 'Support Vector Machine': Pipeline([ ('scaler', StandardScaler()), ('clf', SVC(kernel='rbf', C=1.0, probability=True, random_state=42)) ]), # 省略其他模型定義代碼 'Multilayer Perceptron': Pipeline([ ('scaler', StandardScaler()), ('clf', MLPClassifier(hidden_layer_sizes=(50,), max_iter=1000, random_state=42)) ]) }# 評估指標計算函數(省略詳細代碼)def evaluate_model(model, X_test, y_test): ...# 主程序def main(): # 數據預處理 X_train, y_train, X_test, y_test = preprocess_data('./Discovery Set.xlsx', './Validation Set.xlsx') # 獲取模型集合 models = get_models() # 初始化結果存儲和繪圖 cv_metrics = {} test_metrics = {} plt.figure(figsize=(10, 8)) # 模型訓練與評估(省略循環訓練的核心代碼) for name, model in models.items(): print(f"\n{'=' * 30}\nProcessing model: {name}\n{'=' * 30}") # 交叉驗證和模型訓練 ... # 結果可視化 ... plt.show() # 打印結果(省略詳細代碼) ...if __name__ == "__main__": main()注:上述代碼省略了特徵列定義的詳細代碼、列對齊和數據清洗的詳細代碼、評估指標計算函數的詳細代碼、模型循環訓練的核心代碼,完整代碼可通過交流社羣獲取。
結果對比
已發表研究中表現最佳的模型為SVM,AUROC值為0.751,且優於三位臨牀專家。DeepSeek生成的模型中,多層感知器(MLP)表現最佳,AUROC值為0.744,接近原始模型;但在準確率(0.775 vs 0.812)和F1分數(0.853 vs 0.861)方面略低於原始模型。
頭對頭分析顯示,DeepSeek生成的最優模型AUROC值(0.744)優於重新實現的原始研究模型(0.616),存在較大差異。對比原始研究的模型性能曲線可見,DeepSeek生成的機器學習模型性能大部分優於原始研究方法,僅AdaBoost模型性能較差,可通過優化網格搜索參數進一步提升性能。
原始研究作者提供的模型測試結果如下:
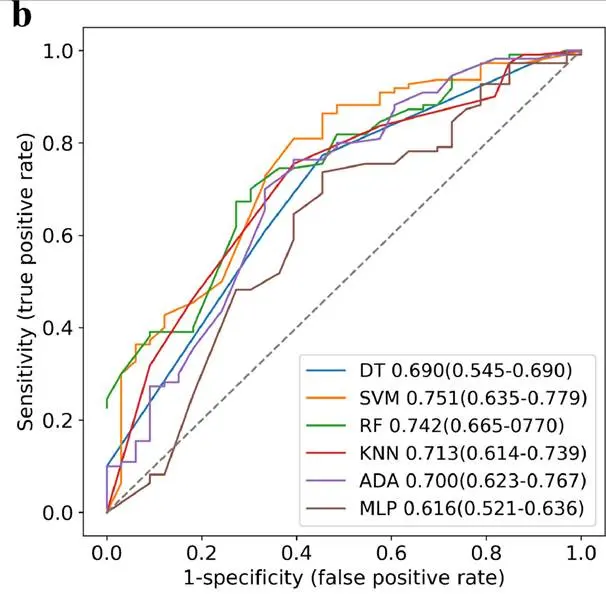
圖2-6 原研究各模型的AUROC值及其特徵曲線
野生型甲狀腺素蛋白澱粉樣變性心肌病的識別
任務説明
轉甲狀腺素蛋白澱粉樣變性心肌病是心力衰竭的常見未識別原因,目前已有針對性治療藥物。因此,在不可逆性心力衰竭發生前,識別高危患者進行早期診斷和治療至關重要。本場景旨在基於健康記錄中的隊列數據和已建立的醫學診斷,構建機器學習模型識別有心臟澱粉樣變性風險的患者。
測試與驗證
基於已發表研究的心臟澱粉樣變性識別任務,提取核心信息輸入"高級Prompt生成器"得到提示詞,通過DeepSeek生成Python分析代碼。模型性能通過AUROC值、準確率和特異度進行評估。
心臟澱粉樣變性識別提示詞的生成過程如下:
圖2-7 心臟澱粉樣變性識別Prompt生成圖
對生成的Python代碼進行文件路徑修改後運行,結果如下表所示:
表2-3 DeepSeek生成的機器模型性能表現
| 機器學習模型 | AUC值 | 準確度 | 特異度 |
|---|---|---|---|
| Logistic Regression | 0.917 | 0.838 | 0.848 |
| XGBoost | 0.948 | 0.886 | 0.925 |
| Random Forest | 0.951 | 0.893 | 0.910 |
各模型在測試集中的AUROC值及其特徵曲線如下:
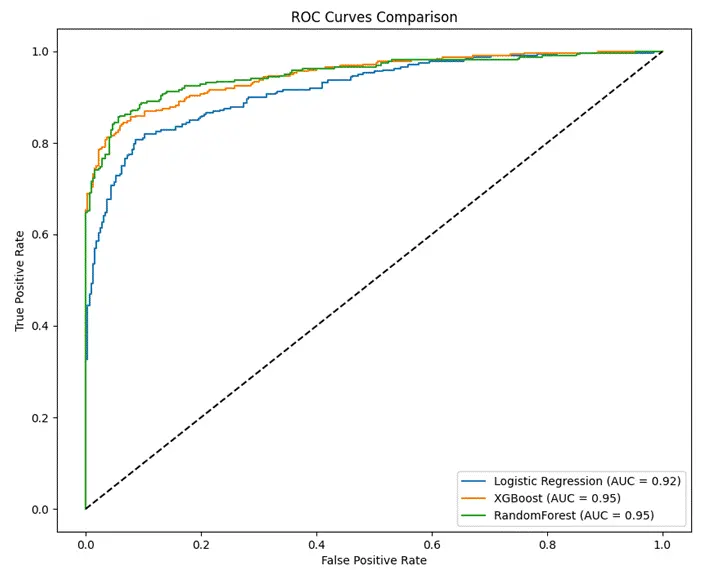
圖2-8 各模型在測試集中的AUROC值及其特徵曲線
核心代碼實現(修改後)
# 導# 數據讀取print("正在讀取數據集...")df = pd.read_csv('./data.csv')# 數據劃分與驗證print("\n=== 數據劃分與驗證 ===")try: # 按隊列類型劃分 train_df = df[df['cohort_type'] == 'Control Group'] test_df = df[df['cohort_type'] == 'Wild Type'] X_train = train_df.drop(columns=['cohort_flag', 'cohort_type']) y_train = train_df['cohort_flag'] X_test = test_df.drop(columns=['cohort_flag', 'cohort_type']) y_test = test_df['cohort_flag'] if len(np.unique(y_train)) < 2: raise ValueError("訓練集需要包含正負樣本")except Exception as e: print(f"\n警告:{str(e)}") print("正在啓用備用劃分方案:分層隨機劃分") # 備用劃分方案(省略詳細代碼) X = df.drop(columns=['cohort_flag', 'cohort_type']) y = df['cohort_flag'] X_train, X_test, y_train, y_test = train_test_split( X, y, test_size=0.3, stratify=y, random_state=42 )注:上述代碼省略了備用數據劃分方案的詳細代碼、模型超參數的完整定義、超參數搜索與模型訓練的核心代碼、模型預測與評估的核心代碼、結果可視化和表格展示的詳細代碼,完整代碼可通過交流社羣獲取。
結果對比
已發表研究中,隨機森林(RF)模型表現最佳,內部驗證集AUROC值為0.930。DeepSeek生成的模型中同樣是RF模型表現最優,且在AUROC值(0.951 vs 0.930)和準確率(89% vs 87%)指標上優於原始模型。
原始研究作者提供的RF模型測試結果如下:
表2-4 原研究提供的RF模型測試結果
| 機器學習模型 | AUC值 | 準確度 | 特異度 |
|---|---|---|---|
| Random Forest | 0.930 | 0.870 | 0.871 |
本章小結
本章通過"高級Prompt生成器"對三類典型臨牀科研場景進行了任務拆解、測試驗證和結果對比。實踐結果表明,該軟件能夠有效實現需求分析中的核心功能,生成的代碼在三類場景中均展現出良好的性能,部分模型表現甚至優於已發表研究的結果,驗證了軟件在臨牀科研輔助中的實際應用價值。
總結
本研究聚焦臨牀科研中工作者面臨的機器學習應用門檻高、代碼實現複雜等核心問題,針對大語言模型在醫學領域應用中存在的提示詞設計專業性不足、輸出質量不穩定等挑戰,提出面向臨牀科研的提示詞工程適配方法,開發"高級Prompt生成器"軟件。
研究採用需求驅動的軟件開發模式,基於PyQt5與Tkinter構建GUI界面,集成數據預處理、模型選擇、評估指標定製等功能模塊;通過思維鏈策略優化提示詞模板,解決模型幻覺問題,並配套開發"醫療數據列名提取系統"提升操作效率。選取三類典型臨牀任務開展驗證,對比DeepSeek生成代碼與原始研究的模型性能,結果顯示生成模型的AUROC值與原始研究差異不顯著,部分場景甚至表現更優,驗證了方法的有效性和可靠性。
技術突破方面,本研究提出醫學領域專用提示詞設計規則,通過角色定義、任務分解與動態約束,使非專業用户可生成高質量代碼,填補了自動化機器學習在自然語言交互方面的空白。實踐意義上,該方案顯著降低了臨牀科研中機器學習的應用門檻,促進了醫學與AI的交叉融合,可有效縮短研究週期。學科推動方面,為醫學大語言模型的應用提供了標準化流程,推動循證醫學任務處理與AI技術的結合,助力精準醫療發展。
本研究仍存在一定不足:一是數據依賴性強,當前模板對數據集結構的預設可能限制泛化能力,未來可引入自適應數據解析算法,支持非結構化醫療數據輸入;二是模型解釋性不足,生成代碼缺乏可解釋性模塊,建議集成模型解釋模板,增強臨牀決策可信度;三是實時性侷限,未考慮在線學習場景。
未來可進一步探索臨牀決策閉環構建,將提示詞生成系統嵌入電子健康記錄平台,形成"問題識別→模型生成→結果反饋"的智能閉環;同時通過構建專業數據庫,讓大語言模型實現自我感知,生成更精準的解決方案。
需要特別強調的是,我們提供24小時響應"代碼運行異常"的應急修復服務,比學生自行調試效率提升40%。我們始終倡導"買代碼不如買明白"的理念,通過人工深度創作降低查重風險,同時保障代碼無漏洞,直擊學生"代碼能運行但怕查重、怕漏洞"的核心痛點。
參考文獻
- 萬豔麗, 王穎帥, 趙姍姍. 醫學大模型研究進展[J]. 醫學研究雜誌, 2024, 53(10): 1-6, 186.
- Singhal K, Azizi S, Tu T, et al. Large Language Models Encode Clinical Knowledge[J]. Nature, 2023, 620(7972): 172-180.
- Gao Y, Xin L, Lin H, et al. Machine Learning-Based Automated Sponge Cytology for Screening of Oesophageal Squamous Cell Carcinoma and Adenocarcinoma of the Oesophagogastric Junction: A Nationwide, Multicohort, Prospective Study[J]. The Lancet Gastroenterology & Hepatology, 2023, 8(5): 432-445.
- Breiman L. Random Forests[J]. Machine Learning, 2001, 45: 5-32.
- Boser B E, Guyon I M, Vapnik V N, et al. A Training Algorithm for Optimal Margin Classifiers[C]. //Proceedings of the 5th Annual Workshop on Computational Learning Theory (COLT). New York, NY, USA: Association for Computing Machinery, 1992: 144-152.
- Luo X, Li F, Xu W, et al. Machine Learning-Based Genetic Diagnosis Models for Hereditary Hearing Loss by the GJB2, SLC26A4 and MT-RNR1 Variants[J]. EBioMedicine, 2021, 69: 103322.
- Huda A, Castaño A, Niyogi A, et al. A Machine Learning Model for Identifying Patients at Risk for Wild-Type Transthyretin Amyloid Cardiomyopathy[J]. Nature Communications, 2021, 12(1): 2725.